여태까지 작은 데이터로 분류했으니 이번엔 약간 조금 더 큰 데이터를 이용해보도록 하겠습니다. 데이터는 여기에서 zoo.csv를 다운 받아주세요. 제가 사용한 데이터의 원문은 여기를 클릭하면 보실 수 있습니다.
DataSet
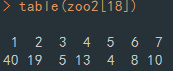
데이터는 동물 종류에 따른 특징들이 있고, 라벨은 포유류, 조류, 파충류, 어류, 양서류, 곤충류, 갑각류 총 7가지가 있습니다.
우선 데이터를 불러와서 동물의 비율이 어떻게 되는지 확인해보겠습니다.
zoo<-read.csv("zoo.csv",stringsAsFactors=FALSE, header =F )
table(zoo[18]) # 종류와 건수 보기
round(prop.table(table(zoo[18])), 2)*100 # 비율 보기
이제 데이터를 정규화하는 작업을 위해 normalize함수를 이번에 만들어보려고 합니다. normalize는 벡터 정규화로써 정규화 방식과 조금 다르지만 knn 알고리즘을 사용할 때 단위를 맞출 수 있는 방법 중 하나입니다. 여기서 TMI로 하나 더 이야기 하자면, 정규화는 데이터 군 내에서 특정 데이터가 가지는 위치를 볼 때 사용하고 표준화는 표준편차를 이용한 식이며 2개 이상의 단위가 다를 때, 대상 데이터를 같은 기준으로 보려고 할 때 주로 사용합니다.
Normalize
우선 normalize 함수를 만들어보도록 하겠습니다.
normalize <- function(x){
return((x-min(x))/(max(x)-min(x)))}
정규화가 잘되는지 확인하기 위해 가라 데이터를 넣어보도록 하겠습니다.
normalize(c(1,2,3,4,5)) # 0.00 0.25 0.50 0.75 1.00
normalize(c(10,20,30,40,50)) # 0.00 0.25 0.50 0.75 1.00
동일한 결과를 얻을 수 있음을 확인 할 수 있습니다. 즉, 정규화가 되엇다는 걸 확인 할 수 있습니다.
이제 데이터 전체에 정규화를 하려고 하는데, normalize 함수에 하나씩 넣을순 없으니, 내장함수 하나와 같이 사용하여 전체 데이터를 정규화 시켜보도록 하겠습니다.
lapply()와 위에서 만들었던 normalize함수를 사용하면 전제 데이터를 정규화 시킬 수 있습니다.
( 단, 라벨 데이터는 정규화 작업을 하시면 안된다는 점 ! )
zoo2_n <- as.data.frame(lapply(zoo[,2:17], normalize))
knn
이제 train과 test 데이터 라벨을 변수로 생성 하겠습니다.
zoo2_n_train <- zoo2_n[1:100, ]
zoo2_n_test <- zoo2_n[101,]
zoo2_train_label <- zoo[1:100,18]
zoo2_test_label <- zoo[101,18]
이제 knn만 돌리는 일만 남았네요. 제 포스팅을 쭉 보셨다면 여태까지 많이 해봐서 익숙하죠 ?
result <- knn(zoo2_n_train, zoo2_n_test, zoo2_train_label, k=1)
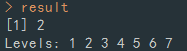
원본 데이터를 확인해보도록 하겠습니다.

마지막 2와 출력 결과인 2가 일치하는 결과를 확인할 수 있습니다.
다음 포스팅에는 유방암 데이터를 이용하여 악성인지 양성인지 분류해보고 조금 더 깊게 들어가기 위해 이원교차표와 적절한 k값을 찾는 방법에 대해 알아보겠습니다. 전체 코드는 여기에서 보실 수 있습니다.
